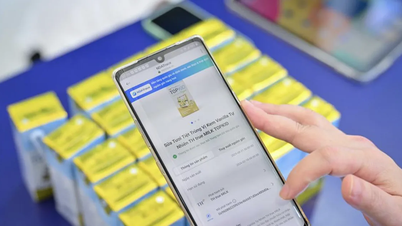
Ein Forschungsteam am Rabin Medical Center (Israel) hat ein bahnbrechendes Modell entwickelt, das die Art und Weise, wie das genetische Risiko für jüdische Paare, die sich auf die Elternschaft vorbereiten, bewertet wird, grundlegend verändern könnte.
Da die Schwellenwerte auf genetisch gemischten Populationen basieren, wie sie in den USA oder Europa vorkommen, kann vielen jüdischen Paaren auf der ganzen Welt bei einem frühen Schwangerschaftsscreening mitgeteilt werden, dass sie „eng verwandt“ sind.
Die Anwendung dieses Standards auf Gemeinschaften mit gemeinsamer Abstammung, wie etwa Juden, Drusen, Tscherkessen oder andere ethnische Minderheiten, führt jedoch häufig zu unnötiger Angst und sogar zu Überlegungen, die Schwangerschaft abzubrechen.
Professor Idit Maya, kommissarische Direktorin des Instituts für Genetik am Rabin Medical Center und Leiterin des Forschungsteams, sagte, ihre Forschung weise auf einen „schwerwiegenden Fehler“ bei der derzeitigen Methode zur Beurteilung des Risikos genetischer Erkrankungen hin.
„Die genetische Nähe jüdischer Paare anhand derselben Kriterien zu messen wie amerikanische Paare, ist ein Fehler“, betont Maya. „In jüdischen Gemeinden, in denen seit Generationen untereinander geheiratet wird, wird ein gewisses Maß an genetischer Ähnlichkeit erwartet. Wird dies nicht berücksichtigt, kann es zu Fehlalarmen und schmerzhaften Entscheidungen wie unnötigen Schwangerschaftsabbrüchen kommen.“
Professor Maya begann ihre Forschung vor mehreren Jahren, nachdem sie wiederholt beobachtet hatte, wie jüdische Paare alarmierende genetische Berichte von ausländischen Kliniken erhielten – in denen die einzigartige genetische Ausstattung der aschkenasischen Juden nicht vollständig verstanden wurde.
Um ihre Hypothese zu testen, tat sie sich mit Professor Lena Sagi-Dain zusammen, der Präsidentin der Israel Society of Medical Genetics und Leiterin der Abteilung für Pränatale Genetik am Carmel Medical Center in Haifa.
Seit 2017 hat das Team im Rabin Medical Center etwa 15.000 genetische Proben gesammelt und analysiert, darunter auch Daten aus Blut und Embryonen.
Die Stichproben wurden nach ethnischer Herkunft klassifiziert – in Israel gibt es etwa 16 verschiedene ethnische Gruppen, darunter Juden aus Regionen wie Buchara, Iran, Kurdistan, Irak, Syrien, Libanon, Libyen, Afghanistan, Jemen und Äthiopien. Sie untersuchten auch arabische Christen, Muslime, Beduinen und Drusen.
Menschliche DNA kommt paarweise vor – eines von jedem Elternteil. Wenn ein DNA-Abschnitt in beiden Kopien identisch ist, spricht man von „Run of Homozygosity“ (ROH). ROH tritt häufig bei Menschen auf, deren Vorfahren in derselben kleinen Gemeinschaft lebten, wodurch sie viele genetische Merkmale teilen.
Das Team maß den Anteil identischer DNA-Segmente in jeder Genomprobe und stellte fest, dass Größe und Häufigkeit dieser Segmente zwischen ethnischen Gruppen stark variierten.
Professor Maya stellte fest, dass die Wahrscheinlichkeit, dass die Eltern einen gemeinsamen Vorfahren hatten, umso höher war, je länger die Überschneidung war – etwas, das in jüdischen Gemeinden üblich ist, wo Mischehen über viele Generationen hinweg vorkommen.
Das Ergebnis ist ein neuer Algorithmus, der die Bewertung des genetischen Krankheitsrisikos anhand des genetischen Profils jeder Bevölkerungsgruppe ermöglicht. Anstatt starrer Schwellenwerte zu verwenden, liefert das Modell „wahre Risikoschwellenwerte“, die auf spezifischen Daten für jede Bevölkerung basieren.
Die aktuellen Richtlinien in den USA und Europa basieren auf Daten von Millionen von Menschen mit unterschiedlichem Hintergrund, was die Bewertungen für genetisch homogene Gemeinschaften wie die Juden manchmal irrelevant macht, so Professor Maya.
Die Israelische Gesellschaft für Medizinische Genetik hat das neue Modell Ende Juli offiziell übernommen und es wird voraussichtlich in die aktualisierten klinischen Leitlinien Israels aufgenommen. Das neue Modell soll unnötige Bedenken reduzieren und „Fehlalarme verhindern“.
Quelle: https://www.vietnamplus.vn/mo-hinh-dot-pha-giup-han-che-canh-bao-di-truyen-sai-trong-thai-ky-post1055197.vnp




![[Foto] Entdecken Sie einzigartige Erlebnisse beim ersten Weltkulturfestival](https://vphoto.vietnam.vn/thumb/1200x675/vietnam/resource/IMAGE/2025/10/11/1760198064937_le-hoi-van-hoa-4199-3623-jpg.webp)

































![[Foto] Der Generalsekretär nimmt an der Parade zur Feier des 80. Jahrestages der Gründung der Koreanischen Arbeiterpartei teil](https://vphoto.vietnam.vn/thumb/1200x675/vietnam/resource/IMAGE/2025/10/11/1760150039564_vna-potal-tong-bi-thu-du-le-duyet-binh-ky-niem-80-nam-thanh-lap-dang-lao-dong-trieu-tien-8331994-jpg.webp)
































































Kommentar (0)